
Strukturelle Biochemie der Meiose
Wie werden homologe Chromosomen während der ersten Reifeteilung (Meiose I) miteinander verbunden? Wie wird die Maschinerie aufgebaut und reguliert?

Jeder von uns hat zwei Kopien von jedem Chromosom, eine von der Mutter und eine vom Vater. Diese Chromosomen sind einander sehr ähnlich und werden als Homologe bezeichnet. Wenn wir Geschlechtszellen (d. h. Spermien oder Eizellen) bilden, müssen wir die Anzahl der Chromosomen in diesen Zellen halbieren, damit unsere Kinder die richtige Anzahl von Chromosomen erben. Dies setzt voraus, dass die Homologe richtig voneinander getrennt werden. Wenn dieser Prozess schief läuft, kann dies zu chromosomalen Störungen wie der Trisomie 21 führen.
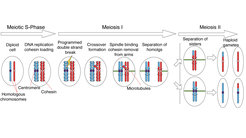
Um voneinander getrennt werden zu können, müssen die Homologen zunächst miteinander verbunden werden. Eine Herausforderung, da homologe Chromosomen normalerweise nicht miteinander verbunden sind. Es gibt einen Mechanismus namens homologe Rekombination (HR), der beschädigte DNA reparieren kann, indem er die korrekte Sequenz vom Homologen kopiert. Während der Meiose macht die Zelle gezielte Doppelstrangbrüche (DSBs) in ihrer eigenen DNA, die dann durch HR repariert werden, was wiederum zu Crossover-Bindungen zwischen den Homologen führt.
Ort, Zeitpunkt und Anzahl der DSBs werden streng kontrolliert, und nicht alle DSBs werden zu Crossovers weiterverarbeitet. Die Homöostase von DNA-Brüchen und Crossovern ist lebenswichtig. Wenn zu viele DSBs auftreten, ist die Integrität des Genoms gefährdet, ebenso wenn DSBs in "verbotenen" Regionen des Genoms entstehen. Bei zu wenigen Brüchen ist es nicht möglich, die für die sichere Verknüpfung von Homologen erforderlichen Kreuzungen zu erzeugen.
Was wir erreichen wollen
Zukünftige Zielsetzungen
Wir wollen unsere biochemischen Rekonstruktionen so weit ausbauen, dass wir im Reagenzglas mit synthetischer DNA eine meiotische Kreuzung erzeugen können. Wir greifen hierzu verstärkt auf hybride strukturbiologische Ansätze zurück und kombinieren verschiedene Methoden, um ein möglichst vollständiges Bild zu erhalten. Ebenso wollen wir über die Verwendung von Hefe als Modellorganismus hinausgehen und beginnen, uns mit Aspekten zu befassen, die für die Meiose von Wirbeltieren und insbesondere des Menschen spezifisch sind. Langfristig hoffen wir, dass unsere Arbeit für Kliniker von Nutzen sein könnte, die Menschen mit Problemen der Fruchtbarkeit oder mit genetischen Krankheiten helfen.
Pressemitteilungen & Neues aus der Forschung








